Nobelpriset i kemi 2002 – Populärvetenskaplig information

English
Swedish
Populärvetenskaplig information
9 oktober 2002
Nobelpriset i kemi 2002 delas mellan forskare som vidareutvecklat två viktiga analysmetoder: masspektrometri (MS) och kärnmagnetisk resonans (NMR). Pristagarna John B. Fenn och Koichi Tanaka (båda för MS) samt Kurt Wüthrich (för NMR) har på olika sätt bidragit till att vidareutveckla dessa metoder till att omfatta biologiska makromolekyler. Detta har inneburit ett revolutionerande genombrott och gjort den kemiska biologin till vår tids ”big science”. Nu kan forskarna snabbt och tillförlitligt identifiera vilka proteiner som finns i ett prov. De kan också få fram tredimensionella bilder av proteinmolekyler i lösning. Forskarna kan alltså ”se” och därmed förstå hur proteinerna fungerar i cellerna.
Revolutionerande analysmetoder för biomolekyler
Varför studera biologiska makromolekyler?
I alla levande organismer – bakterier, växter och djur – är det samma typer av stora molekyler, makromolekyler, som ansvarar för det vi kallar liv. Det som händer i cellerna styrs av nukleinsyror (som DNA) som kan kallas cellernas ”regissörer”, medan de olika proteinerna är cellernas huvudaktörer. Varje protein har en biologisk funktion som kan variera med omgivningen, som t.ex. proteinet hemoglobin som transporterar syre till alla kroppens celler.
Forskarna har länge intresserat sig för proteiner, men ändå är proteomiken, dvs. studier av hur olika proteiner och andra ämnen samverkar i cellen, ett relativt nytt forskningsområde som har vuxit enormt på senare år. I takt med att fler och fler organismers genuppsättning kartläggs flyttas forskningsfronten snabbt framåt samtidigt som nya frågor föds: Hur kan det komma sig att människans ungefär 30.000 gener kodar för hundratusentals olika proteiner? Vad händer om en gen skadas eller saknas helt? Hur uppkommer sjukdomar som t.ex. Alzheimer eller Galna ko-sjukan? Kan den nya kemin användas för att snabbare diagnostisera och behandla de sjukdomar som idag hotar mänskligheten?
För att kunna ägna sig åt frågor som dessa, är forskarna i ständig jakt på mer kunskap om proteinerna och hur de fungerar tillsammans med varandra och med andra molekyler i cellerna. En första förutsättning för denna kunskap är att ta reda på hur proteinerna egentligen ser ut. Det är nämligen små variationer i strukturen som bestämmer proteinets funktion. Nästa steg är att studera dynamiken: Hur ser proteinmolekylerna ut just när de samverkar med varandra och andra molekyler? Vad händer egentligen i de avgörande ögonblicken? Vi behöver se för att förstå.
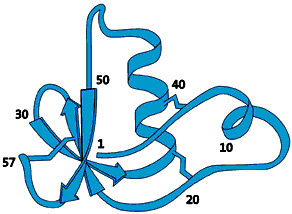
Fig 1. Proteinet består av en lång kedja av aminosyror som veckas, viks och snurras ihop till ett ”nystan”. Det är denna tredimensionella bild av proteinet man vill komma åt för att förstå proteinets funktion. Denna proteinmolekyl, som var en av de första att helt strukturbestämmas med NMR, har en ungefärlig diameter på en miljondelscentimeter (10-8m).
Masspektrometri – en metod att identifiera molekyler
Med masspektrometri kan man idag snabbt identifiera ett ämne i ett prov med avseende på dess massa. Det är en teknik som forskarna länge använt på små och medelstora molekyler. Tack vare metodens känslighet är det möjligt att spåra mycket små mängder av varje molekylslag. Doping- och narkotikatester, livsmedelskontroll och miljöanalyser är exempel på områden där masspektrometri idag används rutinmässigt.
Grunderna för masspektrometrin lades redan i slutet av 1800-talet. De första analyserna av små molekyler rapporterades 1912 av Joseph J. Thomson. Flera av 1900-talets Nobelpris har varit direkt beroende av masspektrometrisk analys. Ett par exempel är Harold Ureys upptäckt av deuterium (Nobelpris i kemi 1934) och upptäckten av fullerener, ”kolfotbollar”, som gav Robert Curl, Sir Harold Kroto och Richard Smalley Nobelpriset i kemi 1996.
Att kunna använda masspektrometri också för makromolekyler var ett mål som länge lockade forskarna. Under 1970-talet gjordes en rad framsteg i att överföra makromolekyler till joner i gasfas, s.k. desorptionsteknik. Detta har legat till grund för den omvälvande utvecklingen av området som vi sett de senaste tjugo åren.
Makromolekyler må vara stora i jämförelse med andra molekyler, men det rör sig ändå om otroligt små strukturer. Hemoglobinmolekylen har till exempel en massa på en tiondels miljarddels miljarddels gram (10-19 g). Hur väger man då något som är så litet? Knepet ligger i att få de enskilda proteinmolekylerna att släppa taget om varandra och sprida sig som ett moln av fritt svävande, elektriskt laddade proteinjoner. En vanlig metod att därefter mäta jonernas massa – och därmed identifiera dem – är att accelerera dem i en vacuumkammare där flygtiden mäts (eng. Time of Flight, TOF). De ”går i mål” i en ordning som dels bestäms av deras laddning, dels deras massa. De snabbaste är de som är lättast och har högst laddning.
Idag finns det två principer att få proteiner att övergå i gasfas utan att tappa sin struktur och form – upptäckarna bakom dessa metoder får i år dela på halva Nobelpriset i kemi. I den ena metoden, där John B. Fenn är upphovsman, sprayar man provet med hjälp av ett starkt elektriskt fält för att åstadkomma laddade, fritt svävande joner. I den andra metoden använder man sig i stället av en intensiv laserpuls. Om detta görs under lämpliga förhållanden (med avseende på energi, struktur och kemisk omgivning i provet) fås en mild överföring av laserpulsens energi till provmolekylerna så att de börjar sväva fritt. Den som först visade att sådan s.k. mjuk laserdesorption kunde användas på stora molekyler som proteiner var Koichi Tanaka.
Fenns insatser – svävning genom sprayning
Under 1988 publicerade John B. Fenn två artiklar som kom att betyda ett genombrott för masspektrometri med ”electrospray” på makromolekyler. I den första studerades polyetylenglykolmolekyler med ökande massa vilket visade att metoden klarar stora molekylmassor med höga laddningar. I den andra publikationen tillämpas metoden också på medelstora hela proteiner. Frisättningen av joner åstadkoms genom att spraya provet med hjälp av ett elektriskt fält så att små laddade droppar bildas. När vattnet successivt avdunstar återstår så småningom de fritt svävande proteinmolekylerna alldeles ”nakna”. Metoden kom att kallas Electrospray Ionisation, ESI.
Genom att molekylerna blir kraftigt positivt laddade, blir kvoten massa/laddning tillräckligt liten för att ämnena ska kunna analyseras i vanliga masspektrometrar. En annan fördel är att en och samma molekyl ger upphov till en serie toppar, eftersom varje molekyl kan ta upp ett varierande antal laddningar. Trots att detta komplicerar mönstret, vilket till en början förvirrade forskarna, innebär det samtidigt information som underlättar identifieringen.
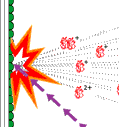 |
Fig 2. Princip för masspektrometri av biomolekyler. |
Tanakas insats – svävning genom sprängning
Samtidigt hände det spännande saker i en annan del av världen. På det japanska instrumentföretaget Shimadzu i Kyoto rapporterade en ung japansk ingenjör om en helt annan teknik för det första kritiska steget; Koichi Tanaka visade vid ett symposium 1987 och ett år senare i en publikation, att jonisation av proteinmolekyler kan uppnås med hjälp av mjuk laserdesorption, SLD (Soft Laser Desorption). En laserpuls träffar provet, som till skillnad från i spraymetoden befinner sig i fast eller trögflytande fas. När provet fångar upp energin från laserpulsen ”sprängs” det i småbitar. Molekylerna släpper taget om varandra och frigörs som intakta svävande molekyljoner med låg laddning, vilka därefter accelereras av ett elektriskt fält och kan detekteras genom att registrera flygtiden. Tanaka var den förste att demonstrera laserteknikens användbarhet på biologiska makromolekyler. Principen är grundläggande för många av dagens kraftfulla laserdesorptionsmetoder, såsom framför allt MALDI (Matrix-Assisted Laser Desorption Ionisation), men också nyare metoder som SELDI (Surface Enhanced Laser Desorption Ionisation) och DIOS (Direct Ionisation on Silicon).
Användningsområden för masspektrometri
Både ESI och SLD är metoder med många användningsområden. De sofistikerade biokemiska analyser man nu kan utföra var för några år sedan bara drömmar. Växelverkningar mellan proteiner är mycket viktiga att studera för att förstå livets signalsystem. Sådana icke-kovalenta komplex mellan biomolekyler kan undersökas med ESI. Metoden är överlägsen andra metoder vad gäller snabbhet, känslighet och identifiering av interaktionen i fråga. Tack vare att de masspektrometriska analysmetoderna är relativt billiga, har de snabbt spridits till laboratorier runt om i världen. Mjuk laserdesorption (i form av MALDI) och elektrospray är idag standardmetoder för strukturanalys av peptider, proteiner och kolhydrater och möjliggör snabb analys av proteininnehållet i hela celler och vävnadsprover. Följande exempel på andra aktuella forskningsområden ger en bild av den mångfald av tillämpningar som årets Nobelpris genererat:
Läkemedelsutveckling
Den tidiga fasen i läkemedelsutveckling har genomgått ett paradigmskifte tack vare ESI med masspektrometri kombinerad med vätskeseparation. Idag kan man med varje instrument rena och studera flera hundra potentiella läkemedelsmolekyler per dag.
Malaria
Forskare har nyligen upptäckt nya möjligheter att tidigt diagnostisera sjukdomen malaria. Genom att analysera malariaparasiterna med mjuk laserdesorption och masspektrometri kan man snabbt bestämma graden av sjukdomens utbredning. Processen förenklas dessutom genom att det mänskliga hemoglobinets syrebärande del, som parasiten tar upp, kan utnyttjas för att ta upp laserpulsenergin.
Äggstocks-, bröst- och prostatacancer
Nya metoder för tidig diagnostik av olika cancerformer har rapporterats i snabb takt det senaste året. Med hjälp av en yta som cancer-specifika biomolekyler fastnar på – och som analyseras med mjuk laserdesorption – kan kemisterna upptäcka cancern tidigare än läkarna.
Livsmedelskontroll
ESI med masspektrometri har också gjort framsteg på små molekyler. De senaste månaderna har det rapporterats att tillagningen av maten vi äter kan ge upphov till en rad hälsofarliga substanser, t.ex. akrylamid, som är en cancerframkallare. Med masspektrometri analyseras snabbt maten under olika stadier av tillagningen och genom modifiering av temperatur och ingredienser undersöker man nu hur de skadliga ämnena kan undvikas eller minimeras.
NMR för biologiska makromolekyler
Om masspektrometrin ger svar på frågor som ”vilket?” och ”hur mycket?” när det gäller t.ex. ett protein, kan NMR sägas ge svar på frågan ”hur ser det ut?”. Även de största proteinerna är alldeles för små för att kunna studeras med tillräcklig upplösning med någon typ av mikroskop. För att kunna göra sig en bild av hur ett protein egentligen ser ut måste man därför använda sig av andra metoder. NMR (Nuclear Magnetic Resonance, dvs. kärnmagnetisk resonans) är en sådan. Genom att tolka topparna i ett NMR-spektrum kan man rita upp en tredimensionell bild av den molekyl man studerar. En finess är att man kan ha provet i lösning, i proteinernas fall deras naturliga miljö i cellen.
Innan NMR fanns att tillgå var röntgenkristallografi den enda metod man hade för att bestämma ämnens tredimensionella strukturer med atomär upplösning. 1957 presenterades den första riktiga tredimensionella strukturen av ett protein, nämligen myoglobin, vilket belönades med ett Nobelpris till Max F. Perutz 1962. Denna metod baserar sig på röntgenstrålars brytning, diffraktion, i proteinkristaller och har sedan dess bidragit till ytterligare en rad Nobelpris. Som ett komplement till röntgenkristallografi sökte forskarna länge efter en metod som också skulle fungera i lösning, dvs. i en miljö som mera liknar den som biomolekylerna naturligt omger sig med.
Fysikerna Felix Bloch och Edward Purcell upptäckte redan 1945 att vissa atomkärnor genom sitt s.k. kärnspinn absorberar radiovågor av en viss frekvens då de placeras i ett starkt magnetfält. Detta belönades med Nobelpriset i fysik 1952. Ett par år tidigare hade man upptäckt att frekvensen för kärnresonans inte bara berodde på magnetfältets styrka och atomslaget, utan även på atomens kemiska omgivning. Dessutom kunde kärnspinnen från olika kärnor påverka varandra vilket gav upphov till finstruktur, dvs. ytterligare ett antal toppar i NMR-spektrum.
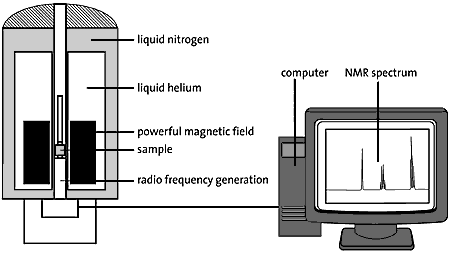
Fig 3. Provet som skall undersökas placeras i ett mycket starkt magnetfält. Här i bilden visas en supraledande magnet, som hålls kyld med flytande kväve och helium. Pulser av radiovågor skickas in i provet, och man avläser provets ”svar” i form av utsända radiovågor. Provsvaret analyseras elektroniskt och behandlas i dator, och resultatet blir ett NMR-spektrum.
NMR-metodens användbarhet begränsades i början av dess låga känslighet: den krävde oerhört koncentrerade lösningar. Men år 1966 påvisade den schweiziske kemisten Richard Ernst (Nobelpris i kemi 1991) att känsligheten kunde ökas dramatiskt om man i stället för att långsamt variera frekvensen utsatte sitt prov för korta och intensiva radiofrekvenspulser. Han utvecklade också, under 1970-talet, ett sätt att avgöra vilka kärnor som låg nära varandra i en molekyl, t.ex. två atomer bundna till varandra. Genom att tolka signalerna i ett NMR-spektrum, kunde man alltså få en uppfattning om molekylens utseende, dess struktur. Metoden var framgångsrik på förhållandevis små molekyler, eftersom man då det gällde större molekyler fick svårt att skilja mellan de olika atomkärnornas resonanser. Ett sådant NMR-spektrum kunde se ut som en gräsmatta i genomskärning – tusentals toppar där man inte kunde avgöra vilken topp som hörde till vilken atom. Den forskare som slutligen löste detta problem var den schweiziske kemisten Kurt Wüthrich.
Kurt Wüthrich – forskaren som visade att NMR för proteiner var möjligt
Kurt Wüthrich utvecklade i början av 1980-talet en idé om hur NMR skulle kunna utvidgas till att omfatta även biologiska molekyler som proteiner. Han uppfann en systematisk metod för att para ihop varje NMR-signal med rätt vätekärna (proton) i makromolekylen (se fig. 4). Metoden kallas sekventiell tillordning och utgör idag en hörnsten i alla strukturundersökningar med NMR. Han visade också hur man därefter kunde bestämma parvisa avstånd mellan ett stort antal vätekärnor, och använda denna information till att med en matematisk metod baserad på distansgeometri beräkna en tredimensionell struktur för molekylen. Den första kompletta proteinstrukturen bestämd med Kurt Wüthrichs metod kom 1985.
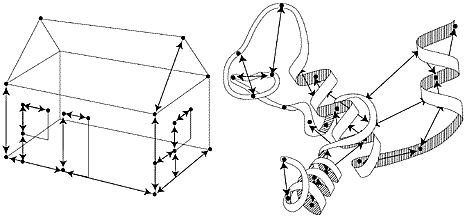 |
| Högupplöst bild (jpeg 168kb) |
| Fig 4. Genom att känna till alla mått på ett hus kan en tredimensionell bild av huset ritas upp. På samma sätt kan man genom att mäta en stor mängd korta avstånd i ett protein bygga upp en tredimensionell bild av strukturen, såsom schematiskt visas i bilden. |
För närvarande är 15-20% av alla tusentals kända proteinstrukturer bestämda med NMR. Övriga är huvudsakligen strukturbestämda med hjälp av röntenkristallografi; några få bestäms med andra metoder såsom elektrondiffraktion eller neutrondiffraktion.
Användningsområden för NMR på makromolekyler
NMR-metoden är i många avseenden komplementär till röntenkristallografi för strukturbestämningar. Om samma protein undersöks med båda metoderna, i det ena fallet i lösning och i det andra kristalliserat, får man i allmänhet samma resultat, med undantag för vissa ytligt belägna delar som påverkas av omgivningen i de båda fallen. I kristallen påverkas ytterdelarna av de tätt packade proteinmolekylerna, i lösning av de omgivande lösningsmedelsmolekylerna. Medan röntgenkristallografins styrka ligger i att noggrant kunna bestämma riktigt stora tredimensionella strukturer, har NMR-metoden andra unika fördelar. Det faktum att undersökningen sker i lösning gör att man kan närma sig fysiologiska förhållanden. En särskild styrka har NMR i att kunna påvisa ostrukturerade och mycket rörliga delar av en molekyl. Man kan klarlägga rörligheten, dynamiken, och hur den varierar längs en proteinkedja. Isotopmärkning kan också användas för att underlätta identifieringen av atomerna.
Ett exempel på NMR-bestämda proteinstrukturer kommer från studier av prionproteiner vilka är inblandade i utvecklingen av en rad farliga sjukdomar som t.ex. Galna ko-sjukan (Nobelpris i medicin till Stanley Prusiner 1997). Här har Kurt Wüthrich med NMR-metodik visat att den friska formen av prionproteiner har två delar: Ungefär halva proteinkedjan antar en välordnad ganska stel tredimensionell struktur i vattenlösning (121-231), medan den andra halvan är strukturlös och mycket rörlig (23-120).
NMR kan även användas för studier av struktur och dynamik i andra biologiska makromolekyler såsom DNA och RNA.
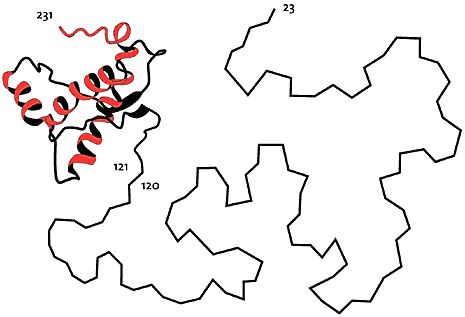
Fig 5. NMR-bestämd struktur av ett prion-protein. Halva proteinkedjan (23-120) är oordnad och mycket rörlig i vattenlösningen.
NMR används också inom läkemedelsindustrin för att bestämma struktur, och därmed egenskaper, hos proteiner och andra makromolekyler som kan vara intressanta målmolekyler för nya läkemedel. Läkemedelsmolekylen utformas så att den passar in i strukturen hos proteinet – som nyckeln i ett lås. Den kanske viktigaste industriella användningen har NMR när man söker efter nya små läkemedelsmolekyler som kan växelverka med en viss biologisk makromolekyl. Om den lilla molekylen binder till den stora, förändras vanligen den stora molekylens NMR-spektrum. Detta kan användas för att ”screena” ett stort antal läkemedelskandidater på ett tidigt stadium av utvecklingen av ett nytt läkemedel.
| Pristagarna | |
| John B. Fenn Virginia Commonwealth University Dept. of Chemistry 1001 W. Main St. P.O. Box 842006 Richmond, VA 23284-2006 USA www.has.vcu.edu/che/people/fenn.html |
Amerikansk medborgare. Född 1917 (85 år) i New York City, USA. Doktorsgrad i kemi 1940 och professor vid Yale University 1967-1987. Professor emeritus 1987 vid Yale University, USA. Professor 1994 vid Virginia Commonwealth University, Richmond, Virginia, USA. |
| Koichi Tanaka Shimadzu Corp. 1. Nishinkokyo Kuwabaracho Nakagyou-ku Kyoto 604-8511 Japan www.shimadzu.com |
Japansk medborgare. Född 1959 (43 år) i Toyama City, Japan. B. Eng. 1983 vid Tohoku University, Japan. Forskningsingenjör vid Life Science Business Unit, Analytical & Measuring Instruments Division, Shimadzu Corp., Kyoto, Japan. |
| Kurt Wüthrich Institut für Molekularbiologie & Biophysik ETH Hönggerberg, HPK CH-8093 Zürich Schweiz och The Scripps Research Institute 10550 North Torrey Pines Rd, La Jolla, CA 92037 USA www.mol.biol.ethz.ch/wuthrich www.scripps.edu/mb/wuthrich/people/kw/kw.html |
Schweizisk medborgare. Född 1938 (64 år) i Aarberg, Schweiz. Doktorsgrad i oorganisk kemi från University of Basel 1964. Professor i biofysik sedan 1980 vid ETH, Zürich, Schweiz. Gästprofessor vid The Scripps Research Institute, La Jolla, Kalifornien, USA. |
Nobel Prizes and laureates
Six prizes were awarded for achievements that have conferred the greatest benefit to humankind. The 14 laureates' work and discoveries range from quantum tunnelling to promoting democratic rights.
See them all presented here.
